第27届国际计算分子生物学大会(Annual International Conference on Research in Computational Molecular Biology,RECOMB 2023)于2023年4月16日到2023年4月19日在土耳其伊斯坦布尔举行。RECOMB作为国际计算生物学领域的顶级会议,吸引了众多国内外专家学者的参与。mgm美高梅79906施建宇教授团队的博士生杜冰雪应邀在大会上进行了题为《MTGL-ADMET: A novel multi-task graph learning framework for ADMET prediction enhanced by status-theory and maximum flow》的口头报告(图1)和海报展示(图2)。在本次会议中,杜冰雪同学荣获了RECOMB 2023授予的Travel Fellowship奖。此外,该论文被收录于RECOMB 2023会议论文集 [1],mgm美高梅79906为该论文的第一完成单位和通讯单位,博士生杜冰雪为该论文的第一作者,mgm美高梅79906施建宇教授和计算机学院于会副教授为该论文的共同通讯作者。


图1 大会论文口头报告现场
图2 海报张贴现场
下文简要介绍了该论文的研究内容。
研究背景:
在药物筛选及设计中,药物代谢动力学及安全问题是导致大部分候选药物失败的原因,且传统的体内和体外的药代动力学及安全性评估方法既耗时又费钱 。吸收、分布、代谢、排泄和毒性(ADMET)是评估类药化合物是一个重要步骤。在早期通过人工智能技术对药物ADMET展开研究,可快速、准确、有效地预测大量化合物的成药性性质,淘汰代谢动力性质不合理的化合物,有效降低新药研发的风险,避免不必要的经济损失。
论文基于状态理论和最大流策略提出了一种新颖的多任务学习框架,即MTGL-ADMET(图3),以筛选有效的正向辅助任务来共同训练目标任务并优化辅助任务的贡献。MTGL-ADMET包括特定任务的分子特征模块、以主要任务为中心的门控模块及任务预测器模块。利用基于有效辅助任务的关系,设计基于多任务的的图神经网络,研究基于吸收、分类、代谢、排泄及毒性预测方法,探索化合物子结构与ADMET的关联规律,它可以促进候选药物筛选或药物设计的发展。
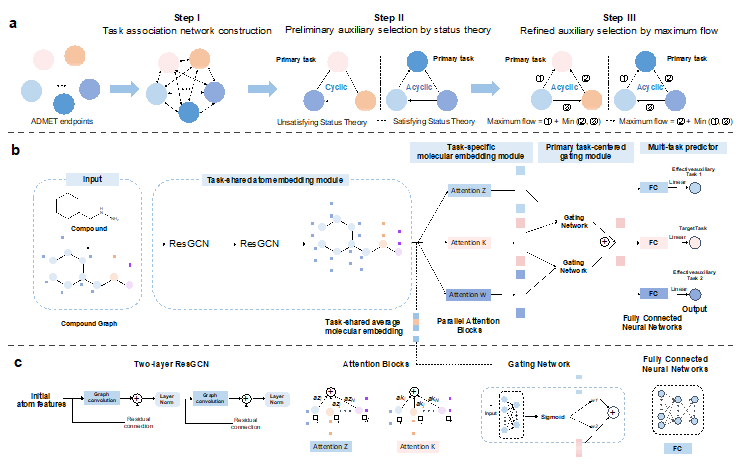
图3 MTGL-ADMET模型框架
参考文献:
[1] Bing-Xue Du, Yi Xu, Siu-Ming Yiu, Hui Yu*, Jian-Yu Shi*. MTGL-ADMET: A Novel Multi-task Graph Learning Framework for ADMET Prediction Enhanced by Status-Theory and Maximum Flow. The 27th Annual International Conference on Research in Computational Molecular Biology (RECOMB 2023). Lecture Notes in Computer Science, pp. 85–103. 文章链接:https://link.springer.com/chapter/10.1007/978-3-031-29119-7_6
(文、图:杜冰雪、施建宇;审核:杨慧)